Join a powerful, unprecedented alliance for better eye health for all.
Join IAPB-
Choose an alternate language here

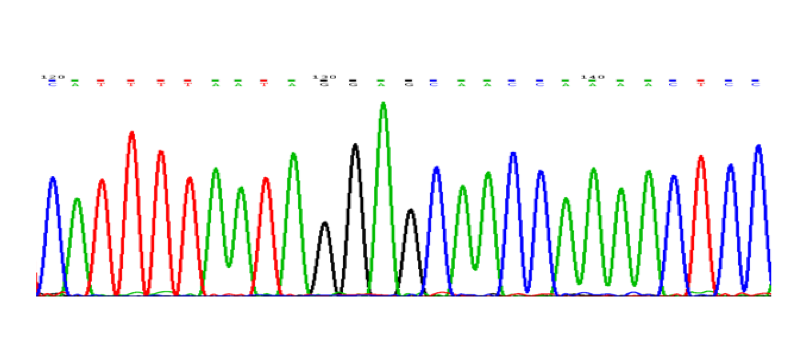
Desde hace más de 40 años, el diagnóstico de una enfermedad se ha realizado con la técnica denominada secuenciación nucleotídica tipo Sanger. Esta técnica es de gran precisión y ampliamente utilizada para el diagnóstico genético en Medicina (Figura 1). Sin embargo, una de sus limitaciones es que permite la caracterización de solamente 600-800 bases de ADN por ensayo, lo que la hace impráctica y costosa cuando el gen a estudiar es muy extenso o en casos en los que la mutación puede suceder en uno de varios genes relacionados y es necesario analizar cada uno hasta identificar la mutación causal. Esta última situación se conoce como heterogeneidad genética (un mismo fenotipo puede sr causado por uno de varios genes).
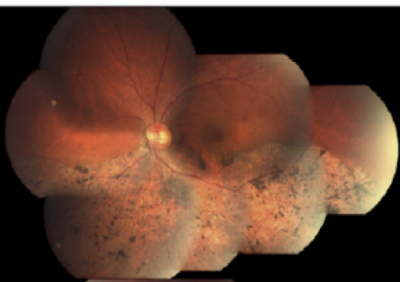
En oftalmología existen numerosos ejemplos de enfermedades monogénicas que presentan heterogeneidad genética, entre las que se incluyen el glaucoma pediátrico, las cataratas congénitas, las malformaciones oculares (microftalmia, anoftalmia y coloboma) y las distrofias retinianas, particularmente la retinosis pigmentaria (Figura 2).
En años recientes se ha desarrollado una nueva tecnología para el análisis del material genético y para la identificación de mutaciones causantes de enfermedades genéticas. Esta tecnología, conocida como secuenciación de siguiente generación (SSG), permite la secuenciación de miles de millones de bases en un solo ensayo. Si se compara esta capacidad con las 800 bases que son posibles analizar con la secuenciación Sanger se comprende por qué la SSG está revolucionando el diagnóstico de las enfermedades genéticas en el humano. Las aplicaciones clínicas de la SSG son muy amplias y actualmente los estudios más utilizados son el análisis de grupos de genes relacionados con alguna enfermedad (secuenciación de paneles de genes) o la secuenciación de la parte de nuestro ADN que codifica para proteínas (secuenciación de exoma). Estos estudios están permitiendo identificar las mutaciones causantes de enfermedades oculares genéticas en un corto tiempo (semanas) y a costos cada vez más accesibles para los pacientes y los servicios de salud. Debe recordarse que la identificación de la mutación responsable de una enfermedad ocular genética tiene beneficios innegables para el paciente y su familia, entre los que destacan la posibilidad de establecer pronósticos, determinar riesgos exactos de repetición de la enfermedad en la familia y conocer si existen tratamientos actuales o futuros que puedan ofrecérsele a los afectados. Un ejemplo claro lo constituye un subtipo de amaurosis congénita de Leber causado por mutaciones en el gen RPE65 para el que ya se dispone de una terapia génica comercial para restauración visual. El análisis de grupos cada vez más numerosos de pacientes con enfermedades oculares genéticas aplicando la SSG está permitiendo conocer además el espectro de mutaciones causantes de estas enfermedades en diversos grupos étnicos.
En México ya se han dado los primeros resultados en enfermedades oculares como la catarata congénita, las malformaciones oculares congénitas y las distrofias retinianas. Un aspecto de gran importancia es que la SSG puede evitar la llamada “odisea diagnóstica” en pacientes con enfermedades oculares de difícil diagnóstico, que consiste en un proceso de años en los que los pacientes son evaluados por numerosos especialistas y sometidos a numerosos estudios sin llegar a un diagnóstico de certeza. La SSG es particularmente útil en enfermedades oculares asociadas a anomalías sistémicas en las que se sospecha un síndrome genético. De este modo, la SSG permite no solo en un mejor abordaje diagnóstico para los pacientes sino también en un ahorro económico considerable para las familias. La oftalmología ha sido una de las primeras especialidades médicas en adoptar esta nueva tecnología de análisis genético como una herramienta de diagnóstico y es previsible que en los próximos años se identifiquen las causas de cientos de enfermedades oculares monogénicas para las que aún no se ha reconocido el gen responsable. La revolución diagnóstica asociada a estos nuevos métodos de análisis genético está permitiendo la consolidación de la llamada oftalmología personalizada, basada cada vez más en las características genéticas de ciertos grupos de pacientes con padecimientos oculares.
Dr. Juan Carlos Zenteno Ruiz
Facultad de Medicina, UNAM-Departamento de Genética, Instituto de Oftalmología “Conde de Valenciana”, Ciudad de México
Photo credit: DennisM2